溶菌酶
| 溶菌酶 | |
|---|---|
 | |
| 溶菌酶的单晶 | |
| 識別 | |
| 符號 | LYZ |
Entrez | 4069 |
HUGO | 6740 |
OMIM | 153450 |
RefSeq | NM_000239 |
UniProt | P61626 |
| 其他資料 | |
EC編號 | 3.2.1.17 |
基因座 | 12 [1] |
| Lysozyme | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 PDB rendering based on 132l. | |||||||||||||
| |||||||||||||
| 标识 | |||||||||||||
| 代号 | LYZ; LZM | ||||||||||||
| 扩展标识 | 遗传学:153450 鼠基因:96897 同源基因:121490 GeneCards: LYZ Gene | ||||||||||||
| EC編號 | 3.2.1.17 | ||||||||||||
| |||||||||||||
| RNA表达模式 | |||||||||||||
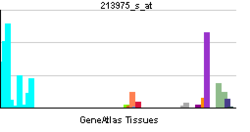 | |||||||||||||
更多表达数据 | |||||||||||||
| 直系同源体 | |||||||||||||
| 物种 | 人类 | 小鼠 | |||||||||||
| Entrez | 4069 | n/a | |||||||||||
| Ensembl | ENSG00000090382 | n/a | |||||||||||
| UniProt | P61626 | n/a | |||||||||||
| mRNA序列 | NM_000239 | n/a | |||||||||||
| 蛋白序列 | NP_000230 | n/a | |||||||||||
| 基因位置 | Chr 12: 69.74 – 69.75 Mb | n/a | |||||||||||
PubMed查询 | [2] | n/a | |||||||||||
溶菌酶(英文名称:Lysozyme,又譯溶解酶)是一个分子量为14.4kDa的酶,它經由催化肽聚糖中N-乙酰胞壁酸和N-乙酰氨基葡萄糖残基间和壳糊精中N-乙酰葡糖胺残基间的1,4-β链的水解,而破坏细菌的细胞壁。一些人体细胞分泌液中含有溶菌酶在,如唾液、眼泪、鼻涕;溶菌酶也存在于粒線體中的细胞质颗粒体和蛋清中。
目录
1 历史
2 生理学功能
3 催化机制
4 相关疾病
5 应用
6 参见
7 参考文献
8 外部链接
历史
1909年Laschtschenko发现蛋清中有杀菌物质。1922年,亚历山大·弗莱明在研究鼻腔粘液的抗菌作用时发现并命名了溶菌酶。[1]
1965年,大卫·菲利浦用X射线衍射技术研究溶菌酶晶体,解析出了2埃分辨率的晶体结构。[2][3]这是第二个使用X射线衍射技术得到的蛋白质结构,也是第一个解析出的酶结构。大卫·菲利浦根据次结构提出了溶菌酶催化的机理,该催化机理最近得到了修正。[4]
生理学功能
溶菌酶是体内免疫系统的一部分,可杀灭革兰氏阳性菌。溶菌酶结合到细菌表面,减少负电荷并协助对细菌的吞噬作用。新生儿缺乏溶菌酶会导致肺支气管发育不良(bronchopulmonary dysplasia)。食用缺乏溶菌酶的配方奶粉会使婴儿腹泻的概率提高三倍。眼泪中缺乏溶菌酶会导致结膜炎。
某些癌细胞会分泌过量的溶菌酶,导致血液中溶菌酶含量过高,造成肾衰竭和低血钾。
催化机制
溶菌酶进攻肽聚糖(细菌细胞壁的组分,特别在革兰氏阳性菌的细胞壁中含量丰富)水解连接N-乙酰胞壁酸和N-乙酰葡糖胺第四位碳原子的糖苷键。整个过程是,首先溶菌酶通过其两个结构域之间的“沟”结合到肽聚糖分子上;随后其底物在酶中形成过渡态的构象。根据Phillips机制,溶菌酶与葡聚六糖结合。然后溶菌酶将葡聚六糖上的第四个糖扭曲为半椅形构象。在这种扭曲状态(能量较高)中,糖苷键很容易就发生断裂。
位于溶菌酶蛋白序列35位的谷氨酸(Glu35)和52位的天冬氨酸(Asp52)的侧链被发现对于溶菌酶的活性非常关键。Glu35作为糖苷键的质子供体,剪切底物的C-O键;而Asp52作为亲核试剂参与生成糖基酶中间体。随后,糖基酶中间体与水分子发生反应,水解生成产物,而酶保持不变。
相关疾病
在一些遗传性淀粉变性症中发现,病人编码的溶菌酶基因中含有一个突变,从而导致溶菌酶在体内多个组织中聚集。[5]
应用
溶菌酶被广泛用于实验室中对细菌所进行的细胞破碎。
由于溶菌酶易于结晶,常被用于各种晶体学相关的研究。
参见
- 肽聚糖
- 糖苷水解酶
- 淀粉变性症
- 细胞破碎
参考文献
^ Fleming A. On a remarkable bacteriolytic element found in tissues and secretions. Proc Roy Soc Ser B 1922;93:306-17
^ Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. Structure of hen egg-white lysozyme. A three-dimensional Fourier synthesis at 2 Ångstrom resolution. Nature, 206, 757-61
^ Johnson LN, Phillips DC. Structure of some crystalline lysozyme-inhibitor complexes determined by X-ray analysis at 6 Ångstrom resolution. Nature, 206, 761-3.
^ Vocadlo, D. J.; Davies, G. J.; Laine, R.; Withers, S. G. Nature 2001, 412, 835.
^ OMIM 105200
外部链接
- 溶菌酶
- 溶菌酶结构和相关文章
| |||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||
